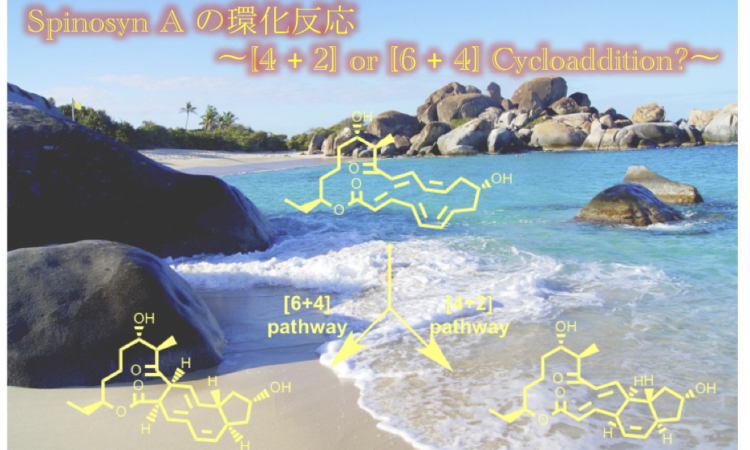
Spinosyn は、バージン諸島に生息する微生物 Saccharopolyspora spinosa から単離された天然物化合物です。その生合成過程に置いて Diels-Alder 反応が進行することでも知られています。
この反応機構については以前から興味が保たれており、すでに 2012 年に Hess らが気相中での QM 計算を行い、協奏的に反応が進むことを報告しています(参考文献 1)。
つい最近、中国の Ruibo Wu のグループが Spinosyn の Diels-Alder 反応の QM/MM MD 計算の論文を ACS Catalysis 出しました。Ruibo Wu と Hess の組み合わせといえば、2015 年に Angewandte に報告されたラノステロールの QM/MM 計算が思い出されます。この二人は仲が良いのでしょうか?Ruibo Wu は質の高い QM/MM 計算の論文をよく発表するため、いつも注目して論文をチェックしています。
Biosynthesis of Spinosyn A: A [4 + 2] or [6 + 4] Cycloaddition?
Nanhao Chen, Fan Zhang, Ruibo Wu, and B. Andes Hess, Jr.
ACS Catal. 2018, 8, 2353−2358. DOI: 10.1021/acscatal.7b03908
概要
Diels-Alder 反応を触媒する酵素の 1 つである SpnF は、spinosyn A の生成に関わっています。これまでの研究で、SpnF は単一の機能をもつ酵素であり、[4 + 2] 付加環化を触媒することが示されています(Keatinge-Clay et al. Nat. Chem. Biol. 2015, 11, 256-258.)。しかし、2016 年の Singleton と Houk の計算 ( J. Am. Chem. Soc. 2016, 138, 3631-3634.) では、[6+4] 付加環化反応が先に起こり、その後 [4 + 2] 付加環化産物ができるのではないかと指摘されおり、SpnF の正確な反応機構については現在でも議論が続いています。本論文では、完全酵素モデルである QM(DFT)/ MM MD シミュレーションを行い、付加環化の方向を制御するために必要な残基、Tyr23、Thr196、および Trp256 を明らかにしました。 これらの残基の作用により、SpnF 内では基質は [6 + 4] の代わりに [4 + 2] の経路を通って進行します。
計算手法
全てのQM/MM 計算、MD 計算は、QChem または Amber を用いて行われました。Houk の先行研究によれば [6+4] 付加環化産物を得るためには分散力の見積りが重要とのことです。そのため、本論文では M06-2X/6-31G(d) または B3LYP/6-31G(d) を用いて計算が行われました。分散関数を使わないのは計算コストのためだと思われます。
内容
2016 年の Houk らの報告では、[6+4] 付加環化反応が先に起こり、その後 [4 + 2] 付加環化産物ができるのではないかと指摘されていますが、計算結果 (27.6 vs 18.6 kcal/mol) が実験結果よりも高すぎることが問題となっています。その一因として考えられるのが、酵素の関与を考慮していないということです。Houk も QM/MM MD 計算を行なっていますが、それは低分子の trajectory 解析であって、酵素を含めてはいません (Houk が Diels Alder 反応の解析をする際によく用いている方法です)。ちなみに、Houk は B3LYP-D2/6-31G(d) で計算しています。
SpnF の X 線結晶構造は 2015 年に報告されています(4PNE)。結晶構造と Docking Simulation の結果を見てみると、活性部位内の残基が付加環化反応に関与しているのではないかという思ったそうです。Tyr23 と Trp256 は ジエンとトリエンの周囲に位置しており、基質と水素結合を作ることが可能な極性残基である Thr196 も反応中心に近い位置にありました。
計算の結果、Tyr23 および Trp256 は C2–C3 二重結合の周りの電子を非局在化し、Thr196 は C13–C14 結合の二重結合特性を低下させることにより、[6+4] の経路は進行しにくくなるそうです。たとえ、[6+4] 中間体ができたとしても、Thr196 により[4+2] の生成物ができます(詳しくは論文を読んでください)。
管理記事中に間違い等ある場合は、コメント欄、twitter またはメールにてお知らせいただけると幸いです。
参考文献
- “The structure of SpnF, a standalone enzyme that catalyzes [4 + 2] cycloaddition”
Christopher D Fage, Eta A Isiorho, Yungnan Liu, Drew T Wagner, Hung-wen Liu & Adrian T Keatinge-Clay
Nat. Chem. Biol. 2015, 11, 256-258. DOI: 10.1038/nchembio.1768 - “Dynamically Complex [6+4] and [4+2] Cycloadditions in the Biosynthesis of Spinosyn A”
Ashay Patel, Zhuo Chen, Zhongyue Yang, Osvaldo Gutiérrez, Hung-wen Liu, K. N. Houk, and Daniel A. Singleton
J. Am. Chem. Soc. 2016, 138, 3631-3634. DOI: 10.1021/jacs.6b00017 - “Concerted, highly asynchronous, enzyme-catalyzed [4 + 2] cycloaddition in the biosynthesis of spinosyn A; computational evidence”
Hess, B. A.; Smentek, L. Org. Biomol. Chem. 2012, 10, 7503. DOI: 10.1039/c2ob25827g
関連する記事
- Coenzyme B12 依存性酵素 ONIOM 計算の汎関数ベンチマーク
- 遷移状態後の枝分かれでの Dynamic Effect
- ダブルハイブリッド密度汎関数理論 doubly hybrid density functional theory
- 正宗・Bergman 環化の計算化学【エンジイン】
- 【スパコン】Titan【アメリカ】
- gaussian scan の結果を解析するプログラム
- 【スパコン】Piz Daint【スイス】
- GRRM の振動計算の結果を Gauss View で解析する方法
- 【スパコン】天河二号【中国】
- GPU を用いた並列計算
- Rh(II) 触媒を用いたインドール合成の計算化学【反応機構解析】
- 構造最適化の閾値は、何を意味しているのか?
- 量子化学計算で a.u. を使う理由 〜Why Atomic Unit?〜