少し時間が経ってしまいましたが、今月初旬にノーベル賞が発表されましたね。
2018 年のノーベル化学賞はファージディスプレイ (Phage Display) と指向性進化法 (Directed Evolution) に授与されました。
今回のノーベル化学賞は計算化学と全く関係ないと思われがちですが、実は David Baker らが取り組んでいる De novo protein (Enzyme) Design と今回ノーベル化学賞の対象となった Directed Evolution は非常に相性が良いことが報告されています。
今回の記事では、De novo Enzyme Design と Directed Evolution を組み合わせた研究の総まとめである、昨年 JACS に掲載された ETH の Don Hilvert の論文を紹介したいと思います。
“Optimization of Enzyme Mechanism along the Evolutionary Trajectory of a Computationally Designed (Retro-)Aldolase”
Cathleen Zeymer, Reinhard Zschoche, and Donald Hilvert
J. Am. Chem. Soc. 2017, 139, 12541−12549. DOI: 10.1021/jacs.7b05796
まずは論文の内容についてを紹介する前に、まずは De novo 酵素デザインと Directed Evolution の二つに簡単に触れたいと思います。
De novo 酵素デザイン
(David Baker, 画像は研究室 HP より転載)
David Baker が取り組んでいる De novo protein (Enzyme) Design は、計算化学の分野においてもっとも注目されている研究課題の一つです。彼のグループは、元々はタンパク質の構造予測に取り組んでいましたが、2010 年前後から De novo 酵素デザインの研究にも取り組み始めました。
De novo 酵素デザインとは、簡単に言ってしまえば、望みの反応を触媒する酵素をコンピューターでゼロから設計してしまおうという試みです。2008 年の Kemp elimination を触媒する酵素(参考文献 3 )、2009 年の Retro-Aldolase(参考文献 4 )、2010 年の Diels-Aldorase(参考文献 5 )の三つは管理人にとっては衝撃的でした。
Diels Alder 反応は、抗体触媒 (catalytic antibody) でも行われていますし、single step の反応ということもあり設計しやすいかと思いますが、retro-aldolase のように複数の TS を経る反応の酵素が設計できるというのは革新的です。
De novo 酵素デザインの大まかな流れは、反応を設計する→量子化学計算で遷移状態を求める→その反応を許容できる大きさのポケットを持つタンパク質を探す→酵素活性部位の最適化→実験による検証、となっております。詳しくは、参考文献 4 の Figure 1 を参照してください。
上述の論文では、Ken Houk が TS を求め、David Baker が活性部位の最適化、Don Hilvert が実験的検証&結晶化、という役割分担がなされていたのではないか管理人は考えています。
Rosetta を用いた enzyme Design については、いずれ記事にしようと思っています。
David Baker は将来ノーベル賞を取るのではないかなと管理人は個人的に思っています。ノーベル賞の受賞に関しては、実用性も一つの大きな指標ですが、管理人の知人達が勤めている複数の会社でも Rosetta や CYRUS BENCH を使っていると言っていましたし、そのうちガンガン工業的にも成果を挙げていきそうな気がしています。
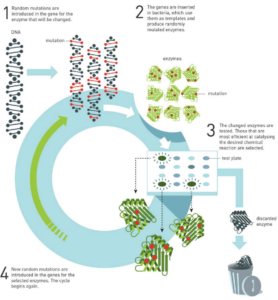
Directed Evolution
(画像は、ノーベル財団の press release より転載)
Directed Evolution については、ノーベル財団がプレスリリースを出していますので、そちらを読むことをお勧めします。PDF リンク
簡単に要約しますと、以下の 1-4 を繰り返し、高活性の酵素を作り出す手法です。
- Error prone PCR などでランダムに変異を導入
- それを大腸菌などに形質転換し、酵素を作らせる
- 望みの機能を持つ酵素をスクリーニングする
- 望みの活性へと変化した酵素の遺伝子情報を調べる
この「突然変異→選択」という過程が自然界での進化に似ているので、Evolution と呼ばれ、さらに、スクリーニング系を変えることにより望みの方向へと進化を促すことができますので、Directed とついています。
大量の変異酵素を効率的にスクリーニングする部分が肝なのですが、Don Hilvert らは、FADS (Fluorescence-activated droplet sorting) と呼ばれるセルソーターのような装置を利用して効率的にスクリーニングしています(参考文献 9)。
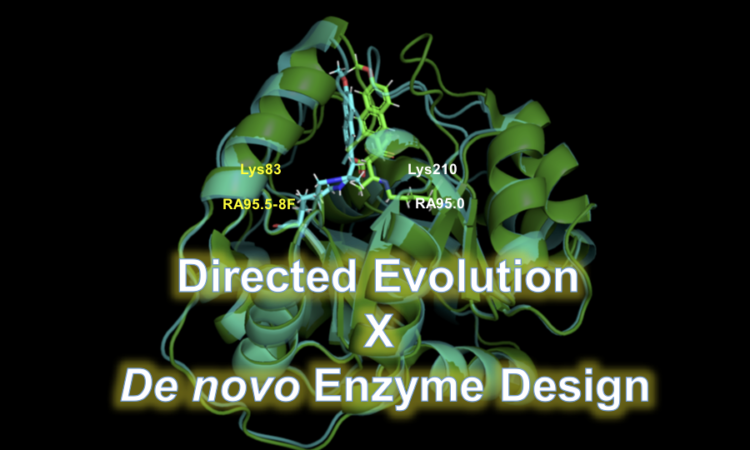
RA95 の進化
(RA95 の触媒する反応スキーム、画像は JACS の論文より転載)
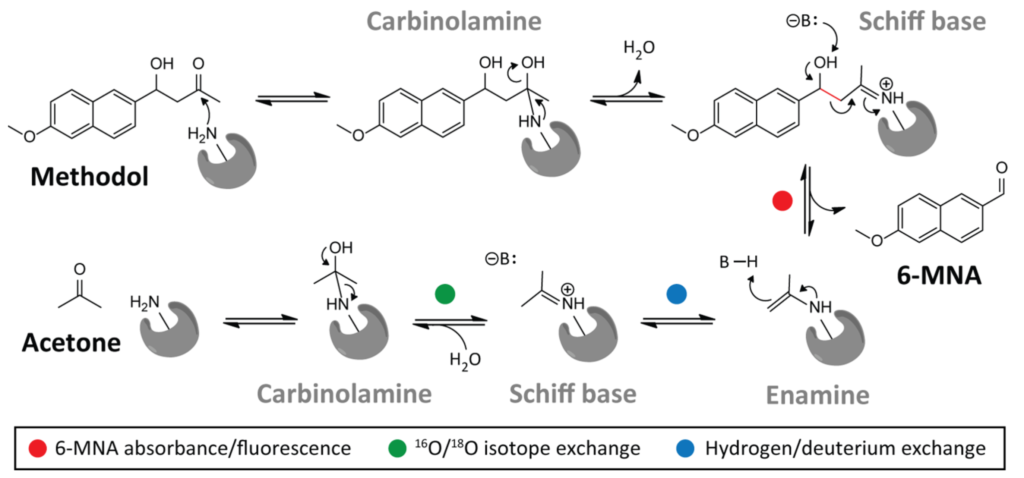
今回取り上げる Don Hilvert の論文では、RA95 という Retro-Aldolase が Directed Evolution によりどのように進化していったかについて論じられています。
この論文を理解するためには、2009 年の Science (参考文献 4 ) 、2012 年の Protein Sci. (参考文献 6)、2013 年の Nature Chemical Biology (参考文献 7)、2014 年の JMB (参考文献 8)、2017 年の Nature Chem. (参考文献 9) の論文をよく理解しておく必要があります。
2009 年の Science の論文で、彼らは retro aldolase の反応機構を 4 種類 (motif) 考え、72 の酵素をデザインしました。そして、32 の酵素において実験的に Retro Aldolase 活性が検出できました。この時、もっとも強い活性を示したものを RA61 と呼びました。また、X 線結晶構造解析により、コンピューターによるモデリングと実際の構造がほぼ同じであることも示しています。
2012 年の Protein Sci. では、さらに 33 個の活性のある Retro-Aldolase を 13 の異なる backbone をもつタンパクを鋳型として作りました。Beta Barrel, Jelly roll, NTF2, Rossman Fold や TIM Barrel などの構造が試されています。 この論文で、TIM Barrel を基に設計した高活性な RA95 が登場しています。今回紹介する JACS 論文の主役です。また、mutagenesis により機能を向上させた RA95.4 も作成されています。
2013 年のNature Chemical Biology では、RA95.5 が得られています。さらに、5 回の Directed evolution を経て得られた RA95.5-5 と計 13 回の directed evolution を経て得られた RA95.5-8 についても詳しく述べられています。
2017 年の Nature Chem の論文では、上述した RA95.5-8 を鋳型としてさらに 6 ラウンドの Direted Evolution を実施し、非常に高活性な RA95.5-8F を取得しています。また、RA95.5-8F は、Aldolase 活性も有しており、広い基質特異性も示しました。
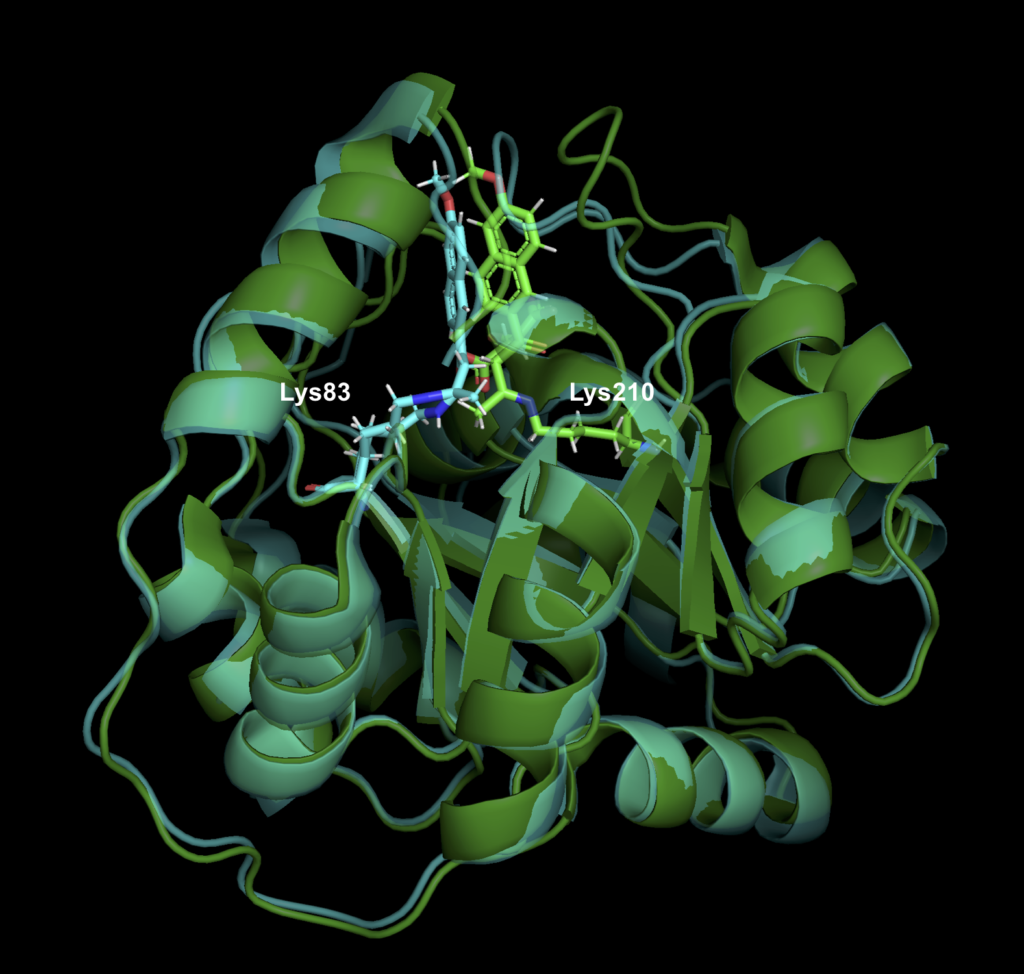
律速段階の変化
(画像は、管理人が Pymol で作成)
当初の設計では、基質と Schiff base を形成するための Lys210 が活性残基でしたが、RA95.5 では Lys83 に変わっています。また、RA95.5-8F では、catalytic tetrad が出現し、Lys83-Tyr-Asn-Tyr で水素結合を形成し、Lys83 のpKa を下げることにより活性を高めています。
さらに、当初の設計では、C-C 結合開裂の段階が律速段階でしたが、Directed Mutation により高活性になった酵素では、最後の product release の部分が律速段階へと変化していました。さらに、天然の Aldolase F6P と比較したところ、こちらも product release が律速段階でした。また、活性部位のメカニズムも非常に似通ったものでした。これは、異なるスタート地点から同じゴールへと至る、酵素の収斂進化を表していると思います。
まとめ
Directed Evolution による進化の過程で、活性残基が変わったり、catalytic tetrad が出現したり、律速段階が変わったり、と酵素化学的に非常に面白いです。特に活性残基が変わるというのは rational な site directed mutation では考えにくいことだと思います。まさに Directed Evolution の強みが出ている感じがします。
今回紹介した論文のように、望みの反応を触媒する酵素の設計は可能となりつつありますが、天然の酵素と同等の高活性な酵素を生み出すことはまだまだ難しいです。しかし、Directed Evolution を組み合わせることにより、望みの反応を触媒する高活性な酵素を得ることが可能となっています。
記事中に間違い等ある場合は、コメント欄、twitter またはメールにてお知らせいただけると幸いです。
参考文献
- David Baker 研究室 HP
- Don Hilvert 研究室 HP
- Röthlisberger, D.; Khersonsky, O.; Wollacott, A. M.; Jiang, L.; DeChancie, J.; Betker, J.; Gallaher, J. L.; Althoff, E. A.; Zanghellini, A.; Dym, O.; Albeck, S.; Houk, K. N.; Tawfik, D. S.; Baker, D. Nature 2008, 453, 190-195. DOI:
- Jiang, L.; Althoff, E. A.; Clemente, F. R.; Doyle, L.; Röthlisberger,
D.; Zanghellini, A.; Gallaher, J. L.; Betker, J. L.; Tanaka, F.; Barbas, C. F., 3rd; Hilvert, D.; Houk, K. N.; Stoddard, B. L.; Baker, D. Science 2008, 319, 1387-1391. DOI: 10.1126/science.1152692 - Siegel, J. B.; Zanghellini, A.; Lovick, H. M.; Kiss, G.; Lambert, A. R.; St Clair, J. L.; Gallaher, J. L.; Hilvert, D.; Gelb, M. H.; Stoddard, B. L.; Houk, K. N.; Michael, F. E.; Baker, D. Science 2010, 329, 309-313. DOI: 10.1126/science.1152692
- Althoff, E. A.; Wang, L.; Jiang, L.; Giger, L.; Lassila, J. K.; Wang, Z.; Smith, M.; Hari, S.; Kast, P.; Herschlag, D.; Hilvert, D.; Baker, D. Protein Sci. 2012, 21, 717-726. DOI: 10.1002/pro.2059
- Giger, L.; Caner, S.; Obexer, R.; Kast, P.; Baker, D.; Ban, N.; Hilvert, D. Nat. Chem. Biol. 2013, 9, 494-498. DOI: 10.1038/nchembio.1276
- Bjelic, S.; Kipnis, Y.; Wang, L.; Pianowski, Z.; Vorobiev, S.; Su, M.; Seetharaman, J.; Xiao, R.; Kornhaber, G.; Hunt, J. F.; Tong, L.; Hilvert, D.; Baker, D. J. Mol. Biol. 2014, 426, 256-271. DOI: 10.1016/j.jmb.2013.10.012
- Obexer, R.; Godina, A.; Garrabou, X.; Mittl, P. R.; Baker, D.; Griffiths, A. D.; Hilvert, D. Nat. Chem. 2017, 9, 50-56. DOI: 10.1038/NCHEM.2596
関連する論文
- もしも計算化学プログラム自体にミスがあったら、、、
- Diels-Alder 反応に最適な汎関数はどれ?
- 歪曲・相互作用モデル distorsion/interaction model
- イオン液体の DMSO 中での酸性度の計算
- AutoDock のスコアファンクションはどれくらい正確か?CASF-2013 ベンチマーク
- 計算化学を用いた天然物の構造訂正
- P450 の量子化学計算【Griseofulvin】
- Spinosyn A の生合成 〜[4 + 2] or [6 + 4] Cycloaddition?〜
- タキサジエン環化反応の分子動力学計算
- 協奏的?それとも段階的? 〜concerted or stepwise?〜
- 【スパコン】神威・太湖之光【中国】
- 【スパコン】天河二号【中国】
- GPU を用いた並列計算
- Rh(II) 触媒を用いたインドール合成の計算化学【反応機構解析】
- 構造最適化の閾値は、何を意味しているのか?
- 量子化学計算で a.u. を使う理由 〜Why Atomic Unit?〜